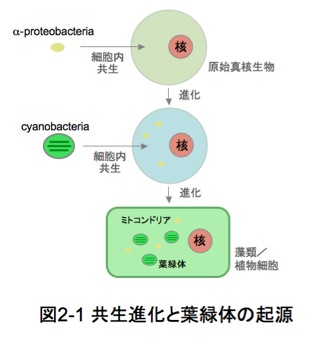
植物細胞には、核とオルガネラ(葉緑体とミトコンドリア)があり、それぞれゲノムDNAを持っていて複製することができます。植物細胞の歴史については、
・研究概要
私たちグループでは、モデル植物として知られるイネ(Oryza sativa)やタバコ(Nicotiana tabacum)を用いて、核コード葉緑体転写因子(PEP, NEP)や核コード葉緑体 鉄-硫黄クラスター転移酵素遺伝子(NifU)の発現と機能解析を行っています。シアノバクテリアや植物葉緑体に存在する普遍の遺伝子発現調節機構を研究することは、光合成効率のアップまたは糖や色素を含めた生物資源の有効活用を通して農業生産に貢献するような優良品種の育種にも役立てられます。
(1) 核コード葉緑体移行型RNAポリメラーゼ(PEP)
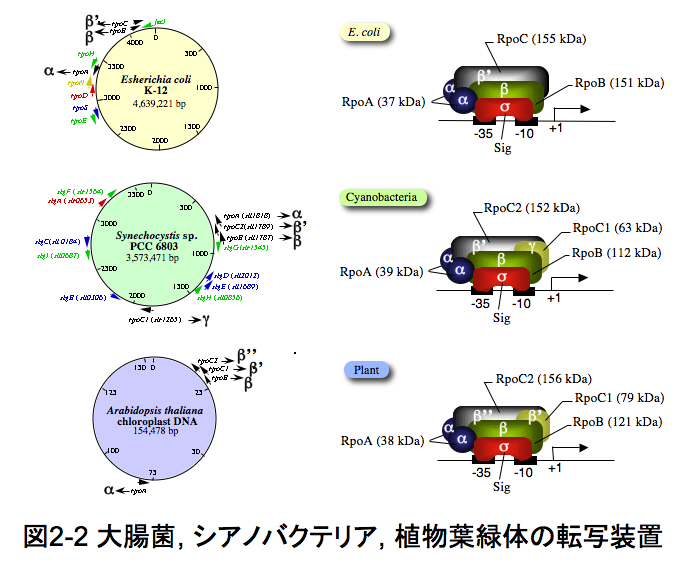
葉緑体の転写装置(RNA polymerase, RNAP)は、シアノバクテリア(原核生物)由来のPEP (Plastid-Encoded plastid RNAP)と呼ばれるものと、ファージ由来のNEP (Nuclear-Encoded plastid RNAP)の少なくても2種類が知られています。PEPは、コア酵素(RpoA, RpoB, RpoC1, RpoC2サブユニットから成る)に「シグマ因子」(Sigサブユニット)が合わさってホロ酵素となり、葉緑体遺伝子プロモーターからの転写を行うと考えられています。シアノバクテリアRNAPと葉緑体PEPの各サブユニットの分子構造は似ています(図2-2)。面白いことに、葉緑体ではコア酵素遺伝子群は葉緑体ゲノム上にコードされていますが、共生進化の過程で、シグマ因子遺伝子は核ゲノム上に吸い取られ、核ゲノム内で転写された後、シグマ因子が葉緑体へ移行し、最終的に葉緑体内で準備されているコア酵素と合体し、RNAPホロ酵素が完成すると考えられています。核コード葉緑体シグマ因子遺伝子は、通常一つの細胞あたり4〜6種類ほど存在していますが、近年、各シグマ因子の発現と機能に関する知見が蓄積されています。
モデル単子葉植物であるイネの場合、核コード葉緑体シグマ因子様遺伝子は全部で6つ同定されていて、OsSIG1, OsSIG2A, OsSIG2B, OsSIG3, OsSIG5, OsSIG6と名付けられました(図2-3C)。
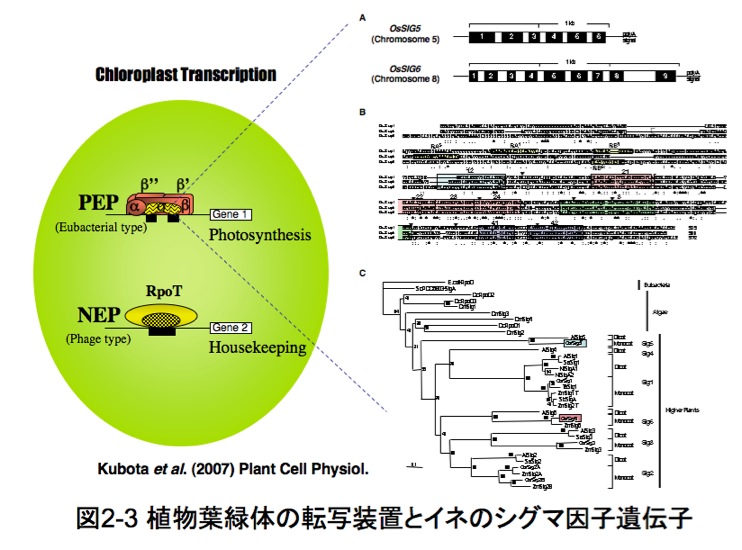
モデル双子葉植物アラビドプシス(Arabidopsis thaliana)に見られるSIG4 タイプは、イネには見あたりません。私たちの研究グループでは、イネの代表品種である日本晴 (Oryza sativa cv. Nipponbare) からOsSIG5とOsSIG6を発見し(図2-3A)、それら遺伝子の発現が光(白/赤/青色光)によって誘導されることを明らかにしました(図2-4)。さらに精製OsSig5蛋白質とシアノバクテリアRNAPコア酵素を再構成させたホロ酵素を準備し、これが葉緑体光合成遺伝子psbA のプロモーターを特異的に認識していることを、in vitro 転写合成系を用い初めて証明しました(図2-4)。モデル単子葉植物におけるSig5タイプのシグマ因子の解析は、これが最初の例となります。
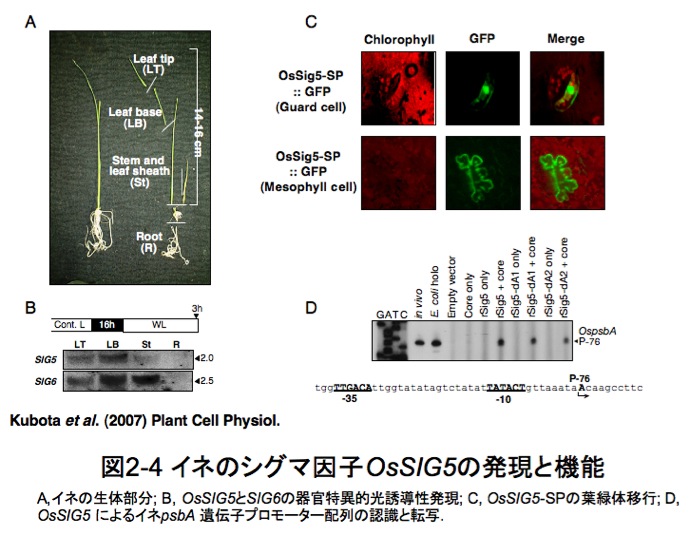
イネpsbA プロモーター配列は、真正細菌の主要シグマ因子が認識する典型的な共通配列 -35(TTGACT) ---17bp--- -10 (TATAAT)と高い類似性を持っていました。 植物葉緑体においてPEPにより認識されるプロモーターは、シアノバクテリアのグループ1(主要シグマ因子)やグループ2型シグマ因子のそれとよく似ています。このことからも、葉緑体とシアノバクリア間における転写装置の機能的なメカニズムは進化的に保存されているということが示唆されます。
こうした保存性をシグマ因子のアミノ酸配列から見ると、シアノバクテリアを含めた真正細菌のシグマ因子と植物葉緑体シグマ因子にはそれぞれ、Region 1, 2, 3, 4という4つの保存領域があります(図2-3B)。私たちは、イネOsSig5とOsSig6のアミノ酸配列を詳細に解析した結果、Region 1の保存領域のさらにアミノ末端側に、植物葉緑体シグマ因子特異的に保存される新規領域を見出し、これをRA/RBと名付けました(図2-3B)。現在、これらRA/RB領域ならびにOsSig5とOsSig6シグマ因子の細胞内での機能を解析中です。
(2) 核コード葉緑体移行型RNAポリメラーゼ(NEP)
葉緑体で機能するRNAポリメラーゼにはPEPとNEPがあることを、先に述べました(図2-3)。NEPはファージ型のRNAPで、基本的に単一触媒サブユニットの酵素だと考えられています。NEP相同性遺伝子はRPOTと呼ばれていますが、RPOT遺伝子は核内で転写された後、サイトゾルで蛋白質として合成され、ミトコンドリアへ移行するタイプ(RpoTm または RpoT1)、ミトコンドリア/葉緑体両方へ移行するタイプ(RpoTmp または RpoT2)、ならびに葉緑体のみへ移行するタイプ(RpoTp または RpoT3)に分けられています。モデル双子葉植物である

NEPとPEPの機能を考えた場合、未分化な色素体 (プロプラスチド)では先ずNEPが活性化され、成熟した葉緑体 (プラスチド)ではPEPが活性化されるというRNAP活性の調節が行われ、これが引き金となって分化に伴う葉緑体遺伝子の発現制御に反映されます。ではNEPの認識する色素体遺伝子プロモーター配列は、PEPのそれと比べてどのように違うのでしょうか? それを調べるためには、色素体でPEPを失活させた植物を作り、NEPにより認識されるプロモーターからの転写を解析する方法が有効です。モデル双子葉植物であるタバコでは、葉緑体形質転換という便利な方法により、葉緑体ゲノム上の標的遺伝子を狙い打って破壊することができます。例えば、PEPを構成する RpoA (或は RpoB, RpoC1やRpoC2)サブユニット遺伝子にスペクチノマイシン耐性遺伝子を挿入し、PEP機能が破壊されたタバコを作ることができます。こうしてできたPEP欠損タバコでは、PEPが認識する光合成遺伝子からの転写ができなくなるので、光合成能の無くなった(葉緑体が未分化なままの)「真っ白」な葉っぱになります。 さらにカルス培地で育てたPEP欠損タバコを野生型タバコに接ぎ木(grafting)すれば、PEP欠損タバコをふやすことができます(図2-5)。このようにして育てたPEP欠損 (NEP活性) タバコを用いて解析すると、葉緑体の遺伝子プロモーターは、(1)PEPのみで認識されているタイプ、(2) PEP/NEP両方で認識されているタイプ、(3) NEPのみで認識されているタイプに分類されました。興味深いことにPEP欠損タバコの未分化な葉緑体では、NEP型プロモーターからの転写が増加します。これらは、NEPとPEPはそれぞれ異なるプロモーター配列を認識しながらも、PEPの機能をNEPが補おうとする姿勢がうかがえます。
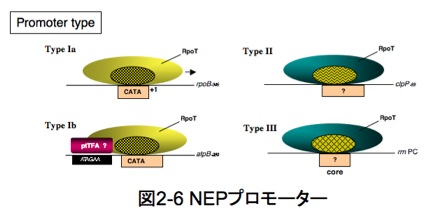
NEP活性を持つ無細胞抽出液を調整し、葉緑体遺伝子プロモーターを鋳型DNAとした in vitro転写実験系を使用した解析が米国や日本ならびに欧州の研究グループからそれぞれ報告されました。その結果、NEP型プロモーターは、Type Ia (CATA配列)、Type Ib (ATAGAA配列 + CATA配列)、ならびにType IIのタイプにクラス分けされ、さらにType IIIタイプも存在することが示唆されました(図2-6)。Type I プロモーターは転写開始点(+1)の上流にCATAという共通コア配列を有していますが、Type II プロモーターではそれが見られず、細胞内では+1よりも下流の配列が転写には必要とされています。
私たちのグループでは、米国ラトガース大学ワックスマン研究所のPal Maliga教授との共同研究で、NEPのType IIプロモーターのコア配列の決定ならびにNEPの機能解析を in vitroとin vivoの両面から行っています。先に述べましたが、双子葉植物(シロイヌナズナやタバコ)色素体で機能すると思われるのはRpoTmpとRpoTpですが、どちらがType IとType II のプロモーターを認識しているのかは明らかになっていません。シロイヌナズナのRpoTpをタバコの葉緑体で発現させると、Type Iプロモーターからの転写が増加することは明らかになっています。シロイヌナズナのRpoTmpをタバコの葉緑体で発現させると、Type IIプロモーターからの転写は増加するのでしょうか? 私たちのグループでは、イネからRpoTmとRpoTpのNEP相同性遺伝子の同定に成功しています。今後は、単子葉植物のRpoTpおよび双子葉植物のRpoTmp/RpoTp遺伝子の発現と機能解析を行っていきたいと思っています。
(3) 核コード葉緑体移行型 鉄-硫黄クラスター因子(NifU)
葉緑体の起源が、シアノバクテリアという原核光合成微生物であることは既に述べました。細胞内共生後、シアノバクテリアの転写制御因子の中には真核光合成生物の細胞内で淘汰され、核ゲノムに移行し定着することなく消滅してしまったものがかなりあります。しかし、シグマ因子などのように葉緑体転写の要として働く遺伝子は、核ゲノム上でコードされるようになり、シアノバクテリアを起源とする転写制御機構は、葉緑体でも継承されています。このように植物葉緑体でも引き継がれ、シアノバクテリアにも存在するような普遍の遺伝子発現調節機構を解明することは、光合成生物の特性の本質に迫る研究として重要です。

モデル植物であるイネの核コード葉緑体移行型遺伝子の機構を解析するには、標的遺伝子の破壊株を取得し、その表現型を解析することが有効です。イネの遺伝子破壊株は、レトロトランスポゾンの一種 Tos17を利用します。Tos17は、野生型のイネゲノム(染色体数12本)上にもともと2コピー存在していますが、イネをカルス培養するとゲノム上に飛散し、コピー数を増やします。その時、たまたま目的遺伝子にTos17が挿入されると、遺伝子破壊株が取得できます(図2-7)。通常は、このようなTos17挿入株をたくさん作製してプールし、それらライブラリーの中から自分達の欲しい遺伝子破壊株をスクリーニングします。そして遺伝子破壊株の表現型を解析することによって、目的遺伝子の機能を明らかにし、それを基に優良品種への改良や育種へと役立てられます。
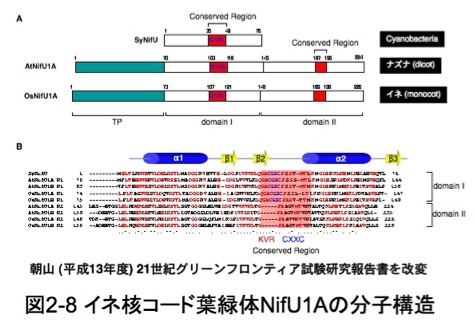
私たちのグループでは、シアノバクテリア PCC6803の約3,000遺伝子とイネTos17挿入破壊の約2万系統の遺伝子とを総当たりで比較し、その中から核コード葉緑体移行型遺伝子の候補となるOsNIFUを見つけ、OsNifU1Aと名付けました。OsNifU1Aは、シアノバクテリアのSsl2667(syNifU)とアミノ酸配列で類似の構造をしています(図2-8)。
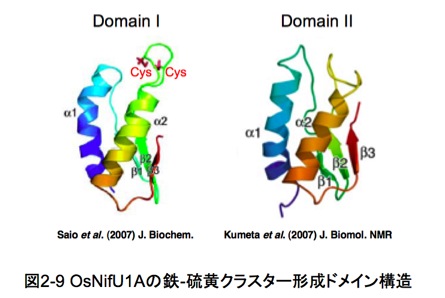
最近、共同研究グループである農業生物資源研究所や北海道大学から、精製OsNifU1A蛋白質立体構造のNMR解析結果が発表されました。それによると OsNifU1Aの domain IIは、α-βサンドイッチ構造をしており、それらは2つのαへリックスがβシートの片側に位置していることが明らかになってきました(図2-9)。Domain IIの構造を基に、domain Iの構造をコンピュータで予測すると、Cys-X-X-Cysモチーフはサンドイッチ構造の中央で、2番目のβシートとαへリックスを結ぶターン部分に位置しているようです。Domain Iで[Fe-S]クラスターを形成した後、これをフェレドキシンへ転移するのにdomain IIが何らかのサポートを効果的にしているのではないかと考えられます。私たちは、現在、OsNifU1Aのトランスポゾン欠損株の表現型を解析すると共に、精製OsNifU1Aを用いた[Fe-S]クラスターのフェレドキシンへの転移実験などを行っています。また、実際にOsNifU1Aが葉緑体へ移行するか調べています。このように、シアノバクテリアと植物葉緑体で共通に機能するモデル遺伝子の発現と産物の機能解析を通じ、光合成生物における普遍の調節機構の一端が解明されることを願っています。